よくある質問
よくあるご質問については以下のページにまとめております
・トラブルシューティングガイド
・解析ソフトダウンロード
・納品レポートの見方
サンガーシーケンス、オリゴ合成
 シーケンス結果を用いて投稿予定ですが必要な情報を教えてください。
シーケンス結果を用いて投稿予定ですが必要な情報を教えてください。
 解析結果グラフにあるPhred Scoreとは何ですか?
解析結果グラフにあるPhred Scoreとは何ですか?
 サンプルおよび解析結果の保存期間は?
サンプルおよび解析結果の保存期間は?
 プライマーはどのように準備して送ればいいのでしょうか?
プライマーはどのように準備して送ればいいのでしょうか?
 DNAはどのように準備した方がいいですか?
DNAはどのように準備した方がいいですか?
 追加反応とは?
追加反応とは?
 再反応を依頼するには?
再反応を依頼するには?
 サンプル受付はいつも可能ですか?
サンプル受付はいつも可能ですか?
 納品(塩基配列結果報告)まで必要な時間は?
納品(塩基配列結果報告)まで必要な時間は?
 サンプル送付において宅配便は指定されていますか?
サンプル送付において宅配便は指定されていますか?
 塩基配列解析を依頼したいのですがどうすればいいですか?
塩基配列解析を依頼したいのですがどうすればいいですか?
- TROUBLE
SHOOTING
GUIDE - ANALYSIS
SOFTWARE
DOWNLOADS
弊社では、初回のシーケンス解析で望ましい結果が得られなかったサンプルにつきまして、弊社の再反応基準の対象となる場合に限り、無料で再反応を承ります。
シーケンシングの失敗は、サンプルの状態や特異的な配列により生じる場合が殆どでございます。このような場合、同一のシーケンシングプロセスにおいて、あらゆる操作を行いましても、改善の可能性がないため、再反応の対象から外れます。 こちらに、頻繁に見られる特徴的な配列の例を掲載いたしましたので、ご参考にして頂けますと幸いです。
マクロジェン・ジャパンの再反応対応につきまして
弊社のシーケンシングサービスにおける再反応は、機械トラブルや研究スタッフのミスの可能性に対する確認作業の一環として行われるもののため、改善の可能性があると判断されるサンプルに限り実施いたします。
そのため、同じ名前のサンプルでありましても、再度送っていただくサンプルは再反応の対象にはならず、解析料金が発生いたしますこと、ご了承ください。
以下に記載の例につきましては、恐縮では御座いますが、ご請求をさせて頂くことを原則としております。
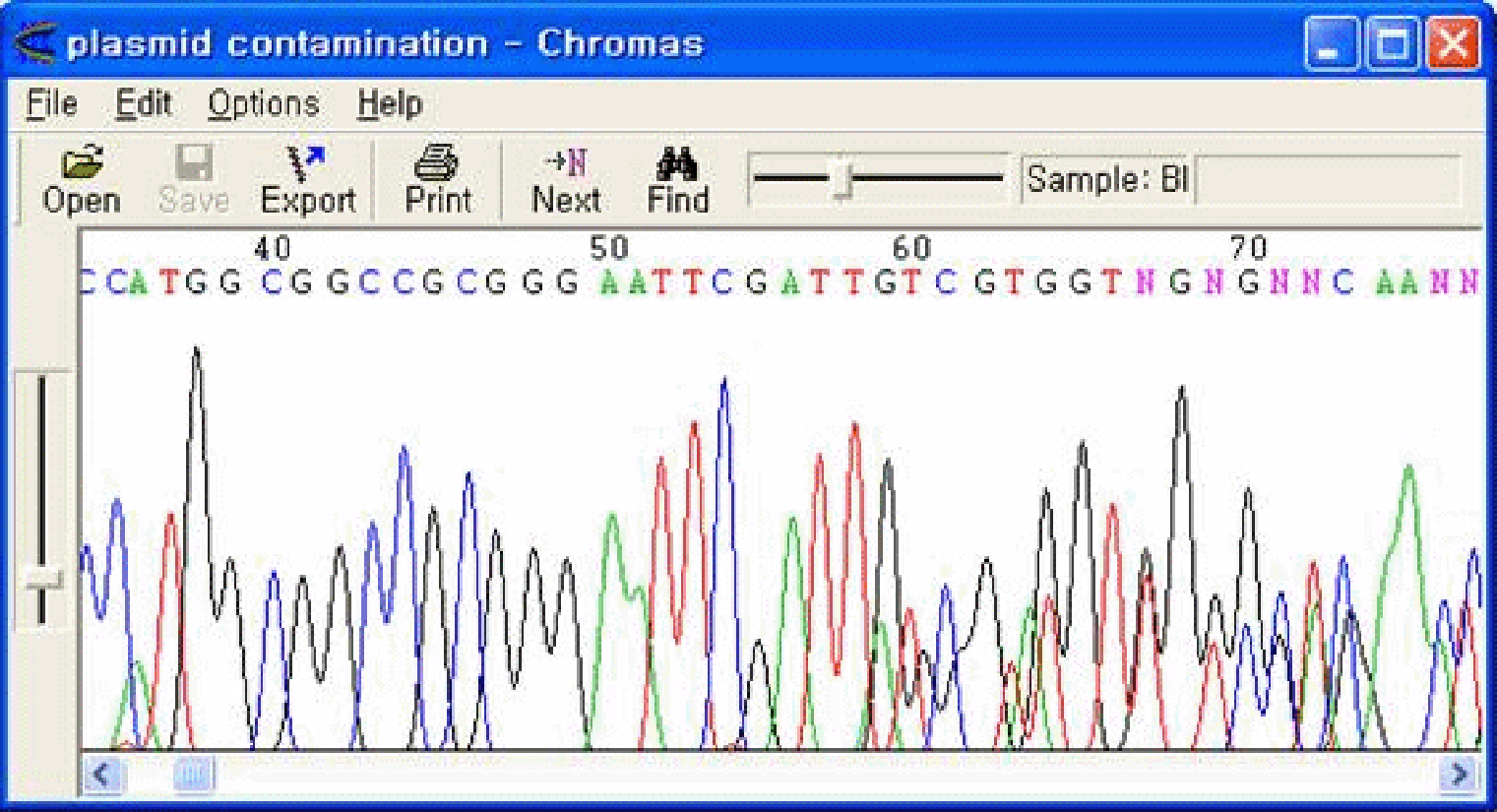
Plasmid DNAをご用意頂く際に、1つ以上のコロニーが同時にpickingされた場合や、1つのコロニーが2つ以上のplasmidを含む場合に、それぞれ異なるinsertが含まれるため、シーケンシングを行うと、ベクターの領域は綺麗なsingle peakを示しますが、insertの領域はmulti peakを示します。
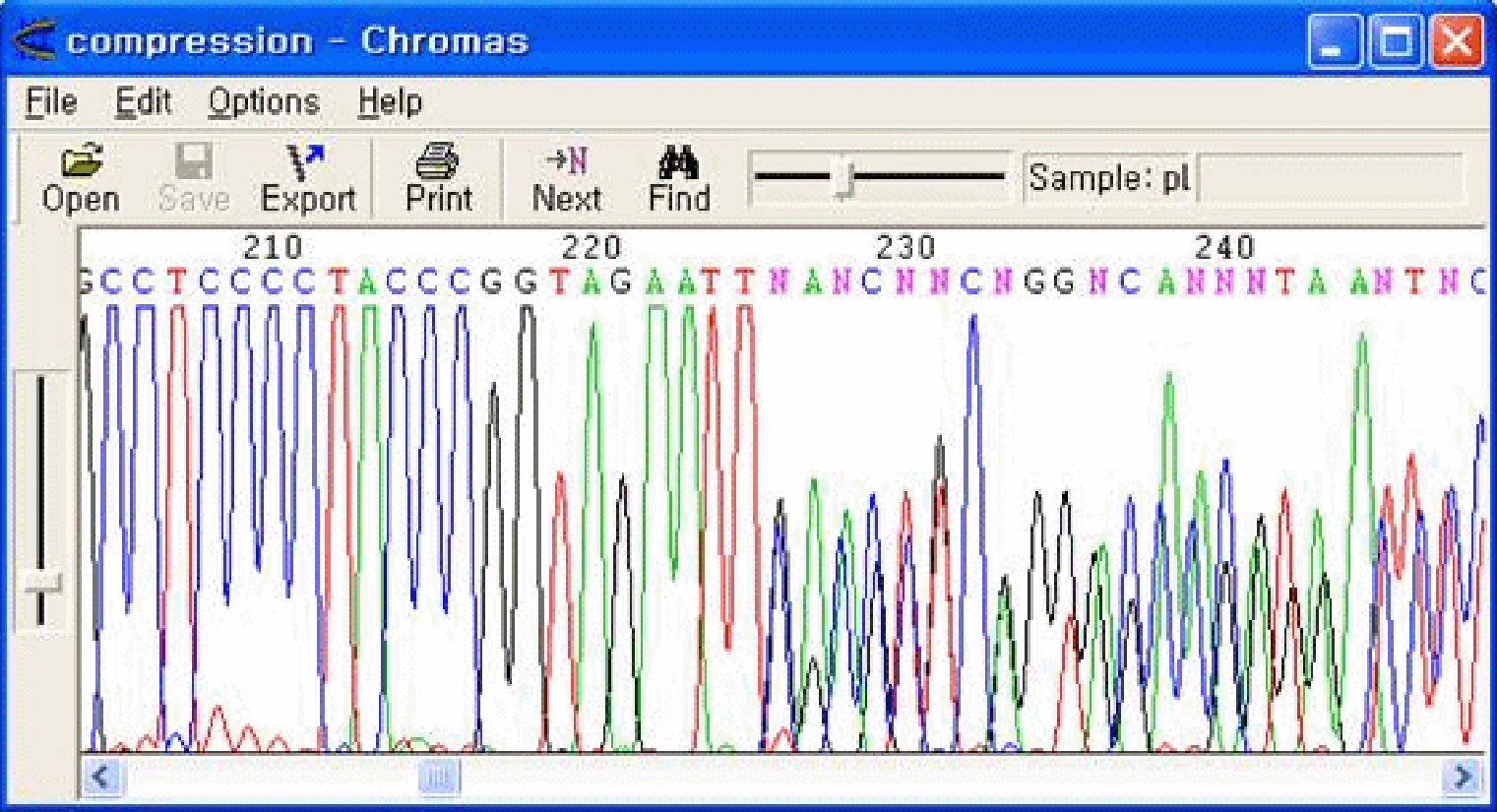
特定の地点からmulti peakが現れる現象は、Mixed Template (insert contamination)と類似しておりますが、multi peakが現れる領域がinsert領域ではない点が異なります。主に二次構造により、同一electrophoretic mobilityからそれぞれ異なるサイズのfragmentを形成することになり、ある地点からことなったpeakが同時に検出される現象です。頻繁ではありませんが、high G/C content, high A/T contentの領域でこのような現象が起きることもあります。
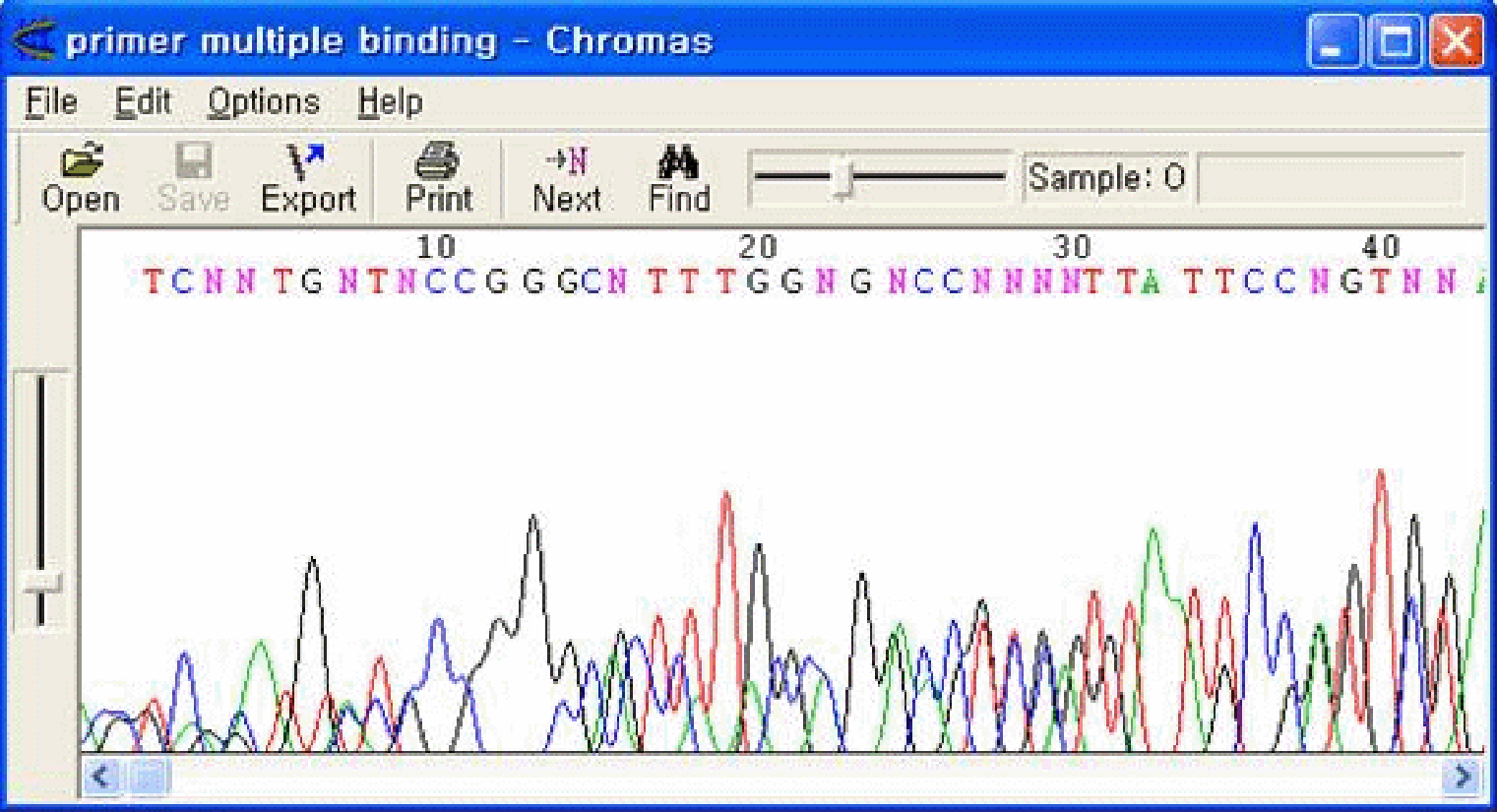
良好なシグナル強度を保ちながら、相互に異なるpeakが共存する現象です。異なる2つ以上のテンプレートが混在しているか、1つのテンプレートであっても、偶然2ヶ所以上でhomologyを持つ場合、プライマーがmulti bindingするため、複数のpeakが現れます。
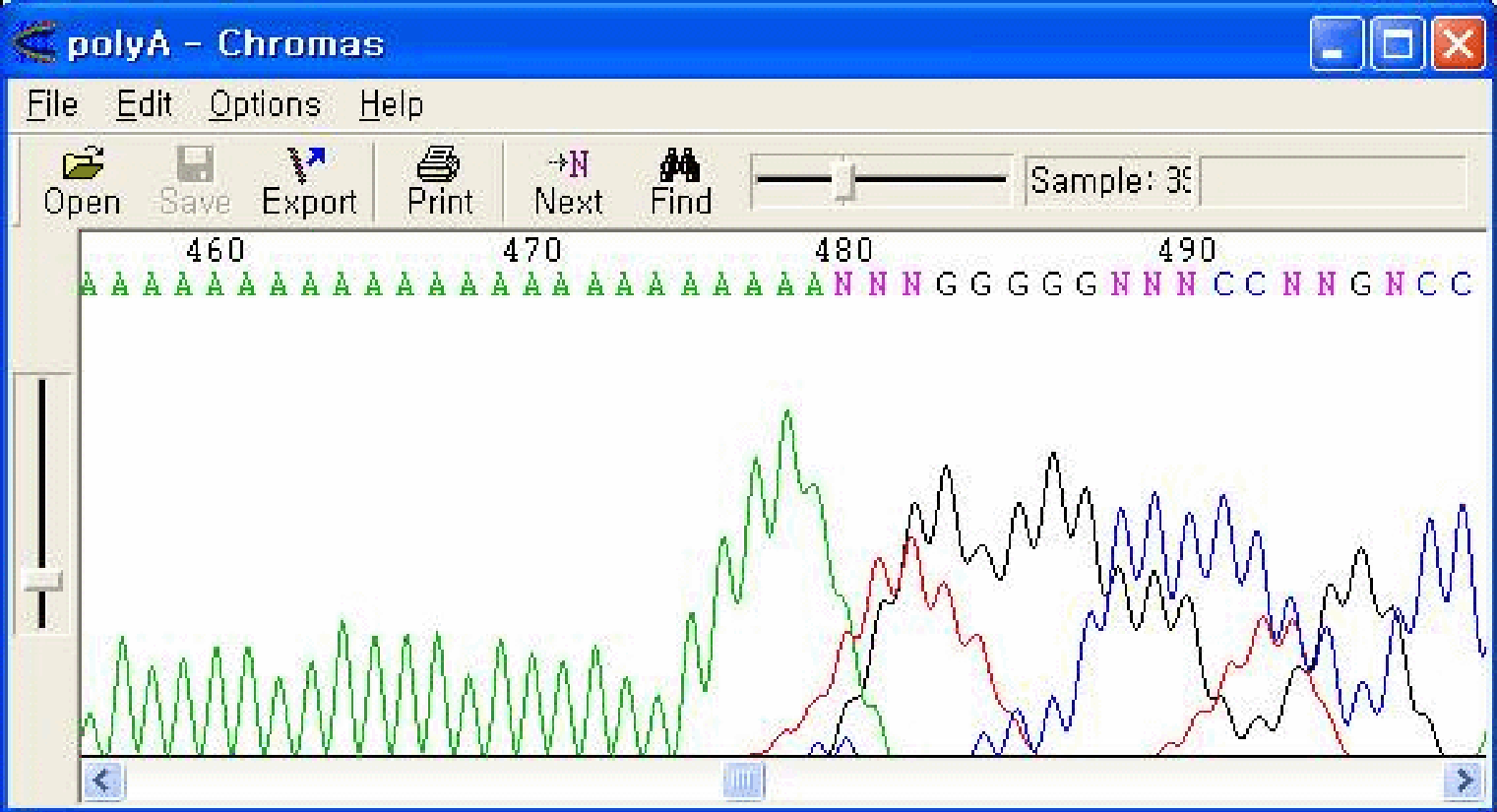
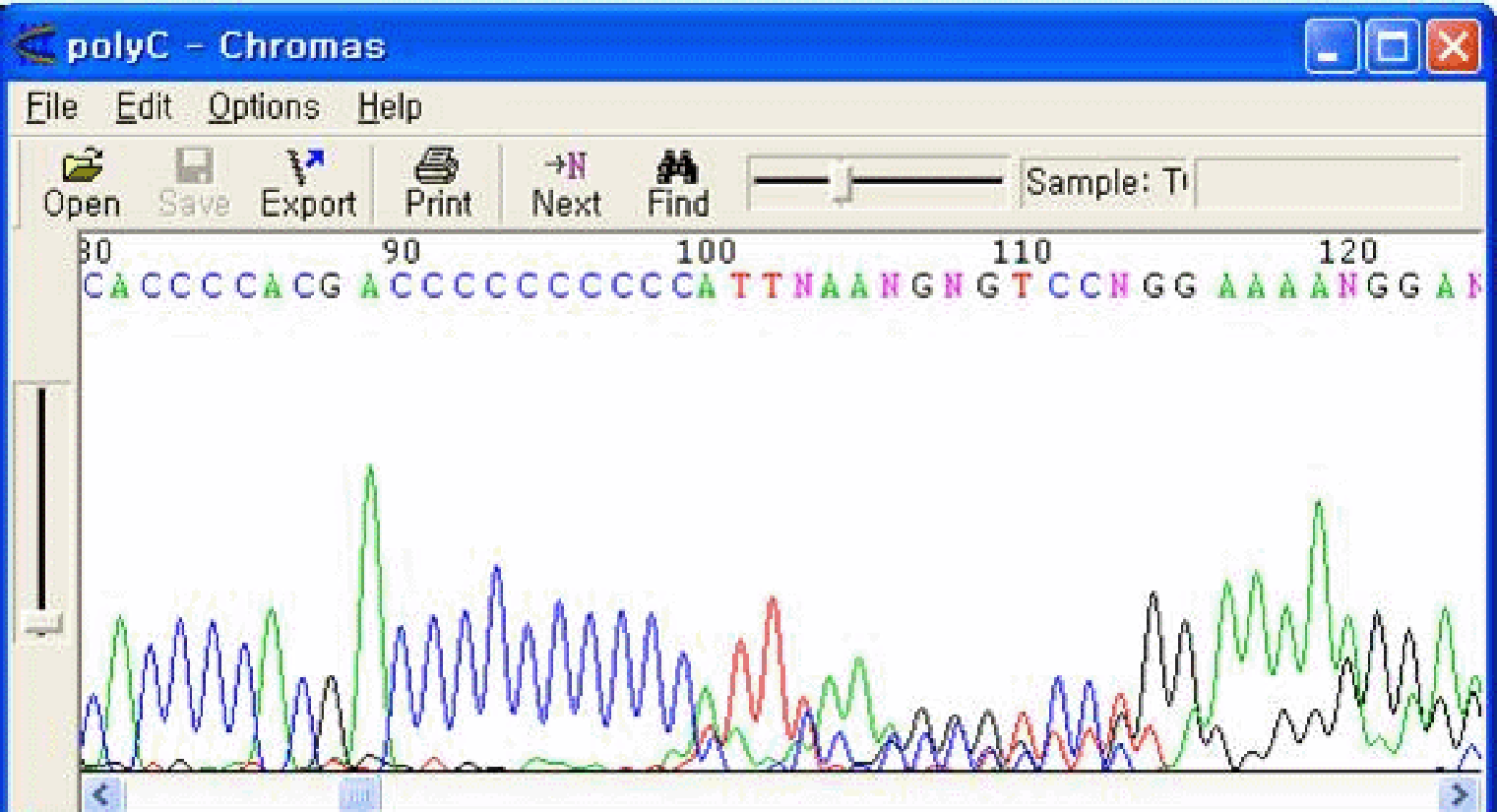
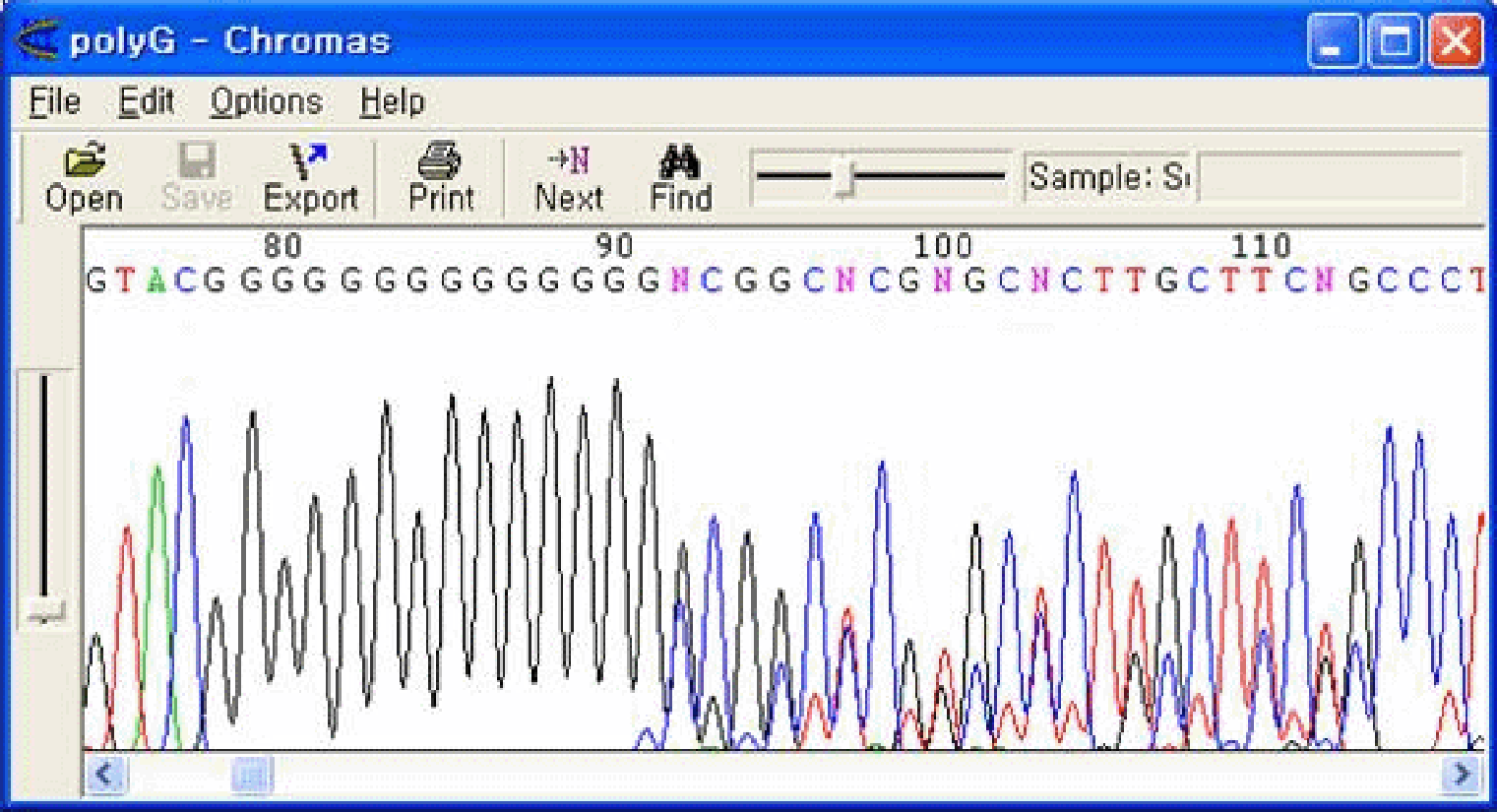
Long homopolymer regionの後でmulti peakが現れる現象です。
Polymerizationする間にhomopolymer regionが正しくpairingすることなく、後ろのbase callingがずれる状態です。
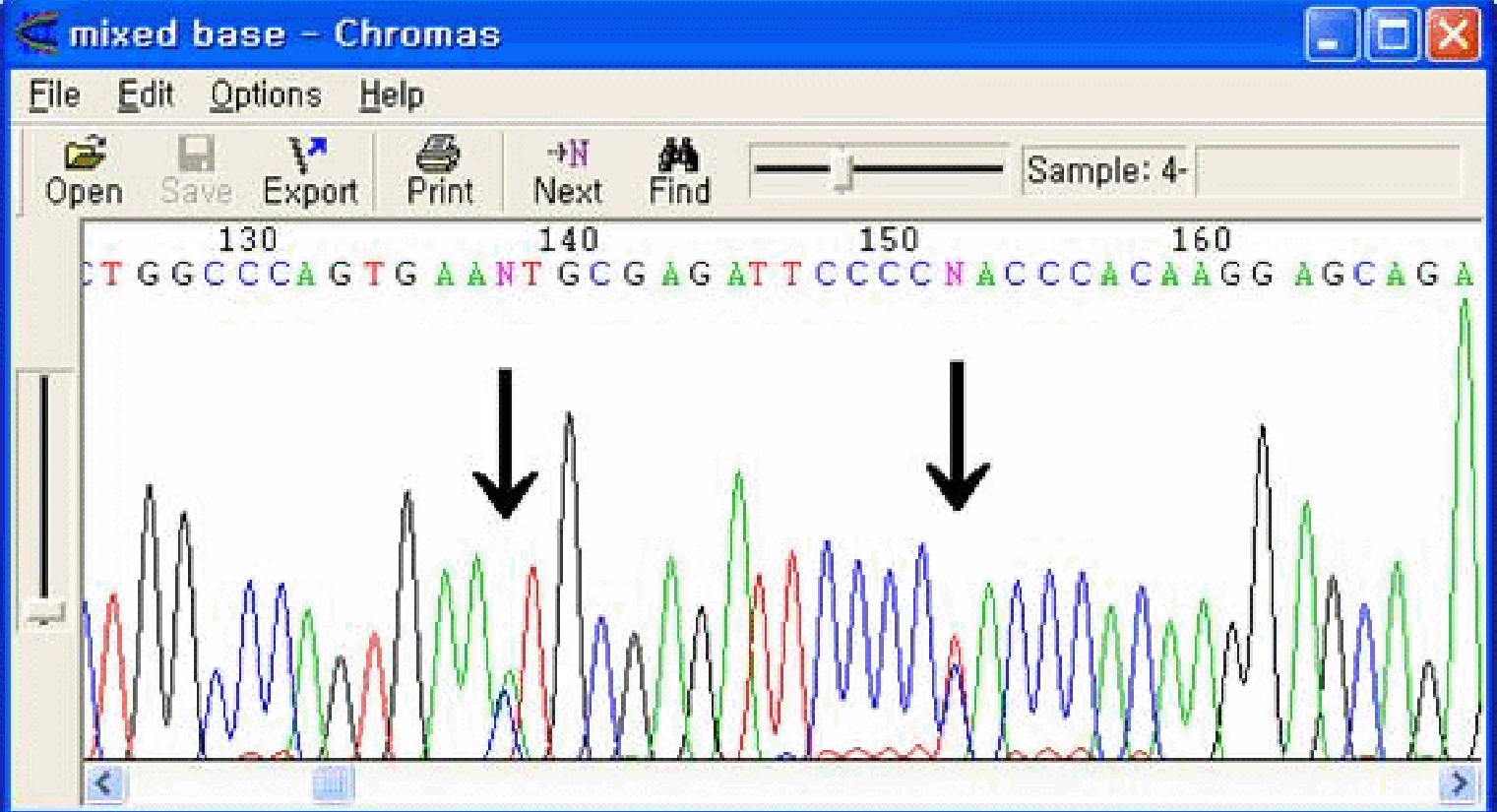
良好なシーケンスデータのうち、部分的に1 baseがdouble peakを示す状態です。SNPの可能性や、頻度は低いですが、point mutationの場合もこのような傾向を示すこともあります。
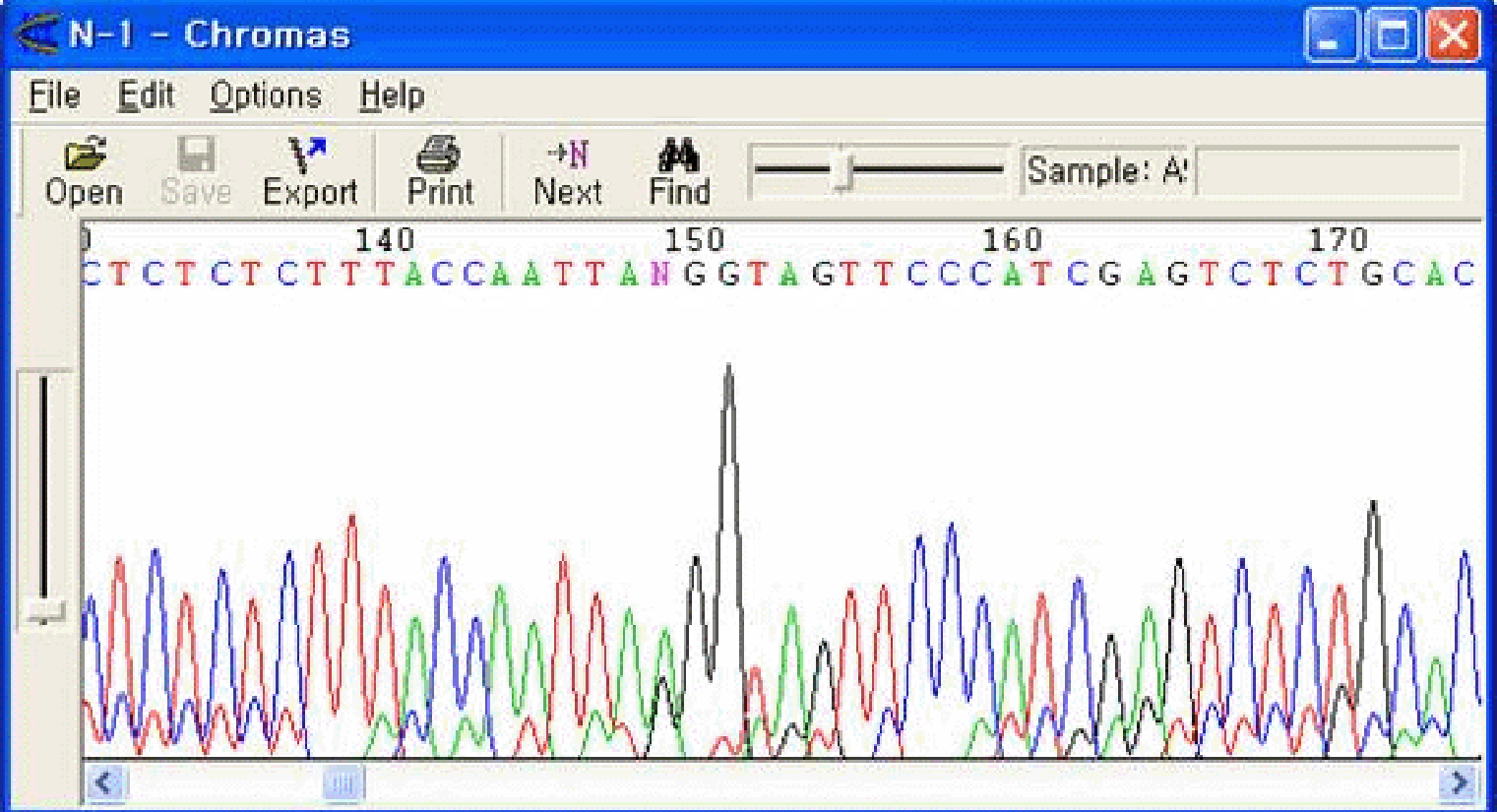
Major peakの下に、同程度あるいは少し低いピークが影のようにバックグラウンドを形成する現象です。これは、プライマーが合成される過程で精製が充分でなかったり、分解を起こした場合に現れます。このような場合は、プライマーを合成し直す必要があります。
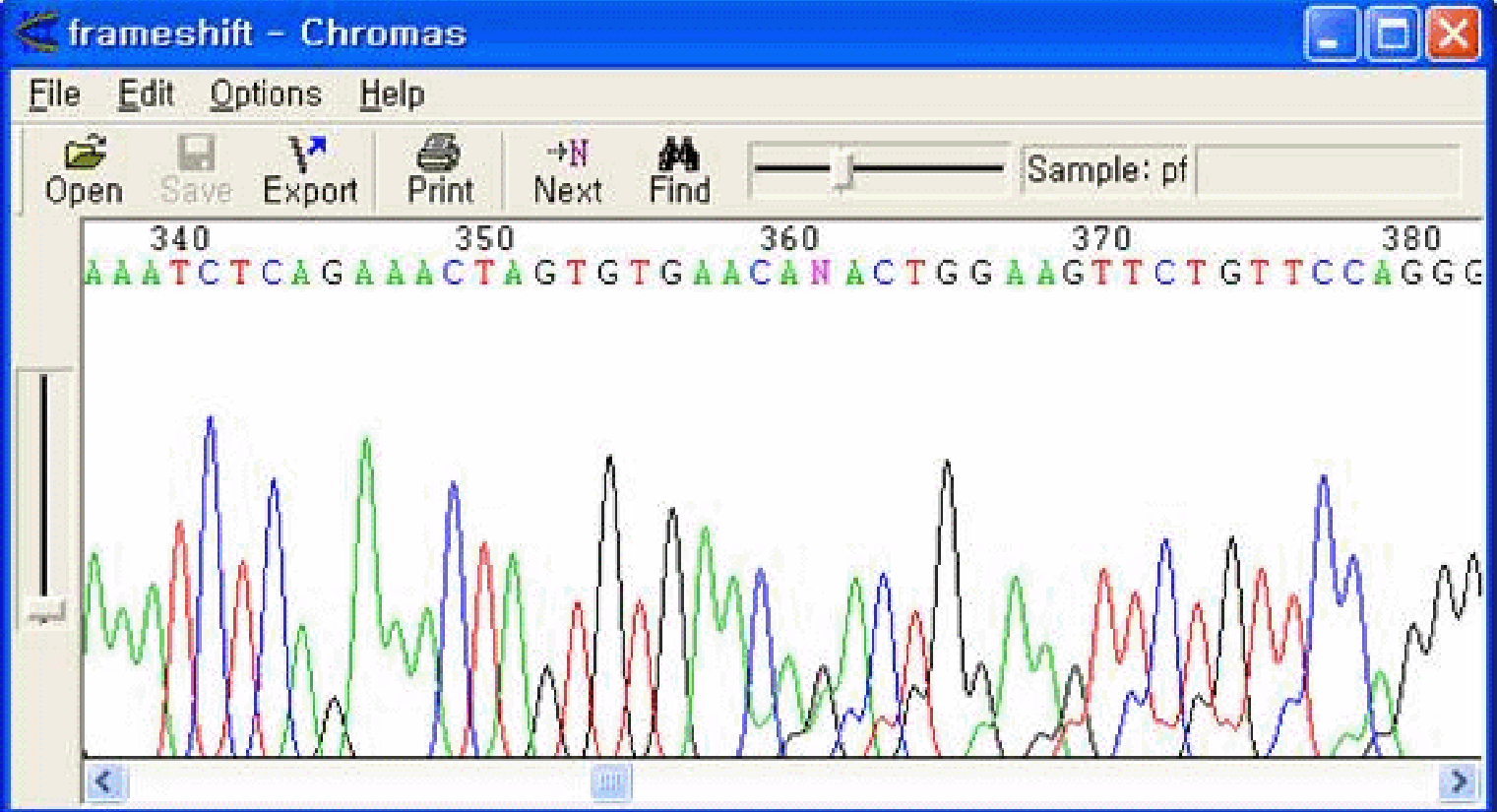
PCR産物において、特定地点からN-1 Primerと同様のminor peakが現れる状態です。N-1 Primerの場合、プライマーの結合した領域直後からminor peakが現れている点が異なります。
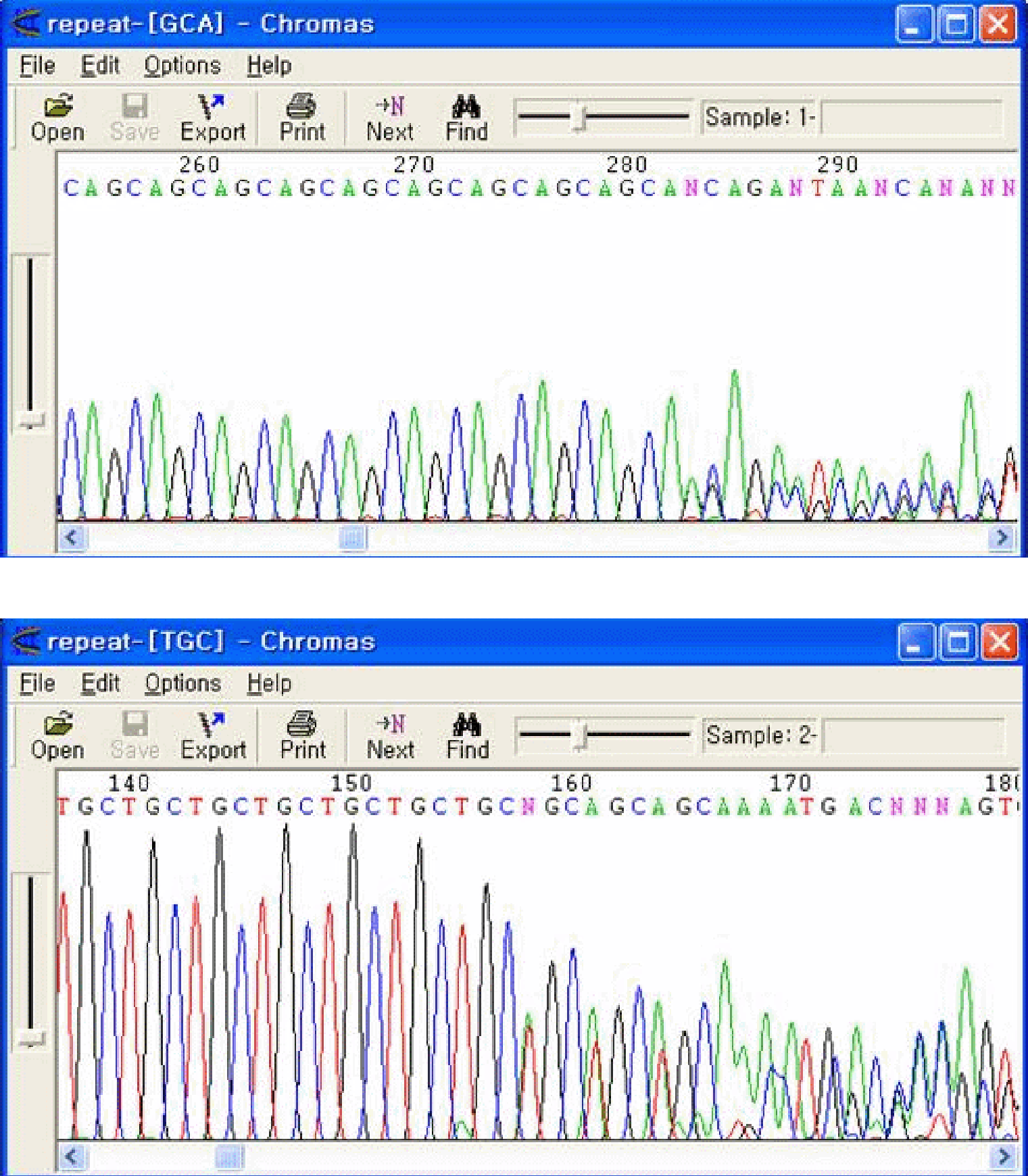
2個あるいはそれ以上の塩基の繰り返し配列が見られ、それ以降にmulti peakを示す現象です。これは繰り返し配列によってそれ以降のポリメラーゼ工程が円滑に進まなくなるためか、二次構造などの形成によるものと報告されています。
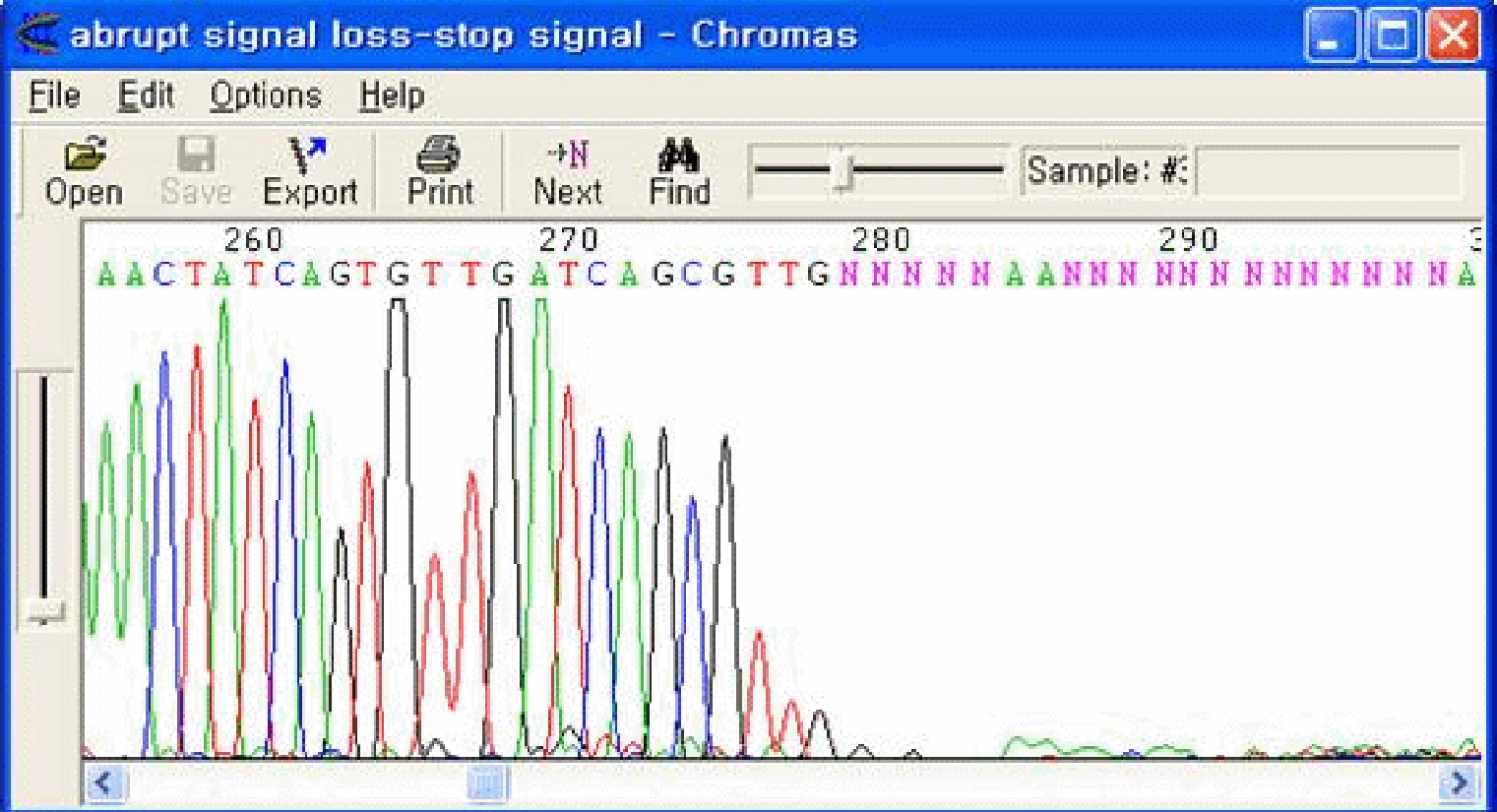
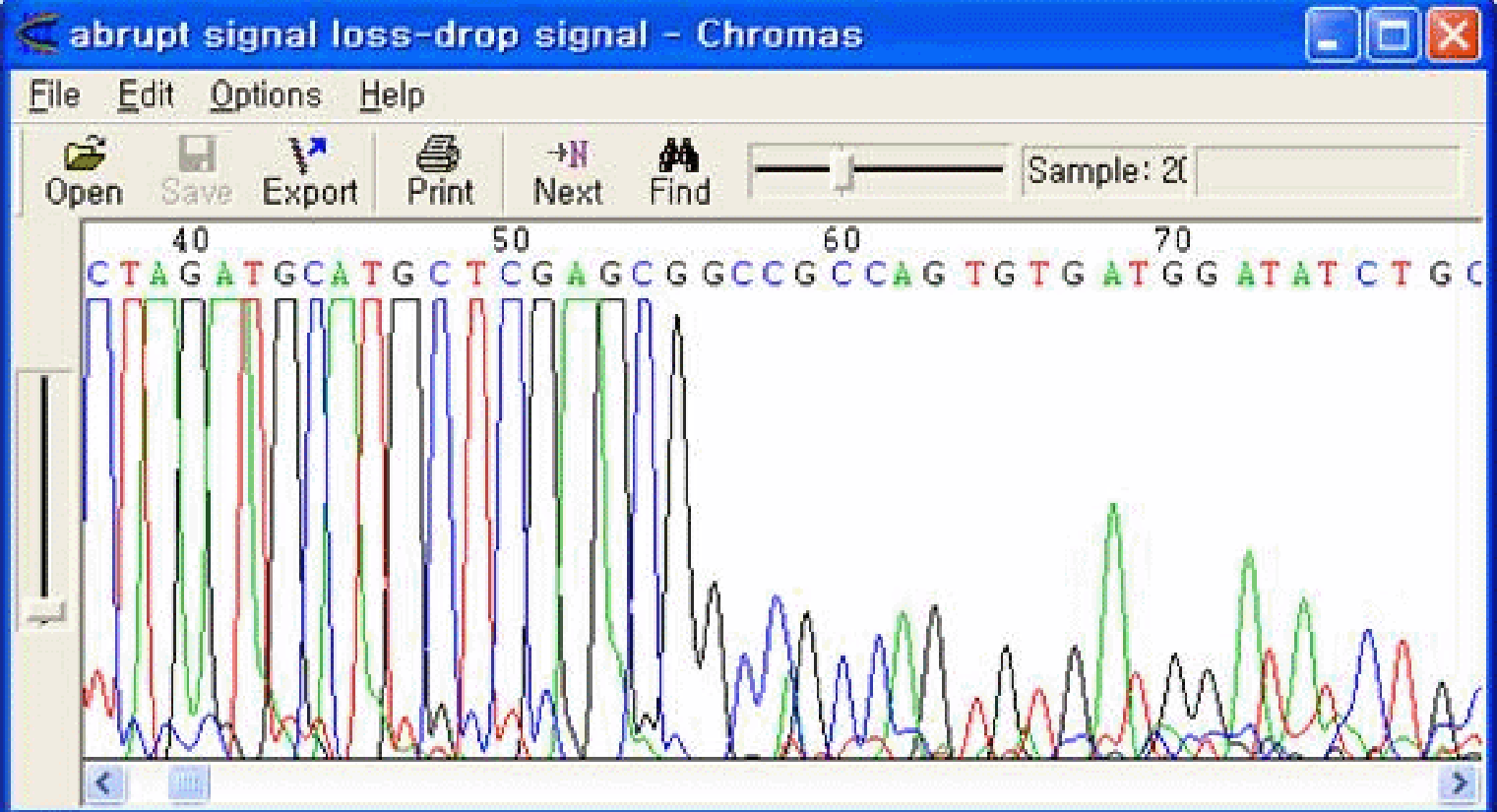
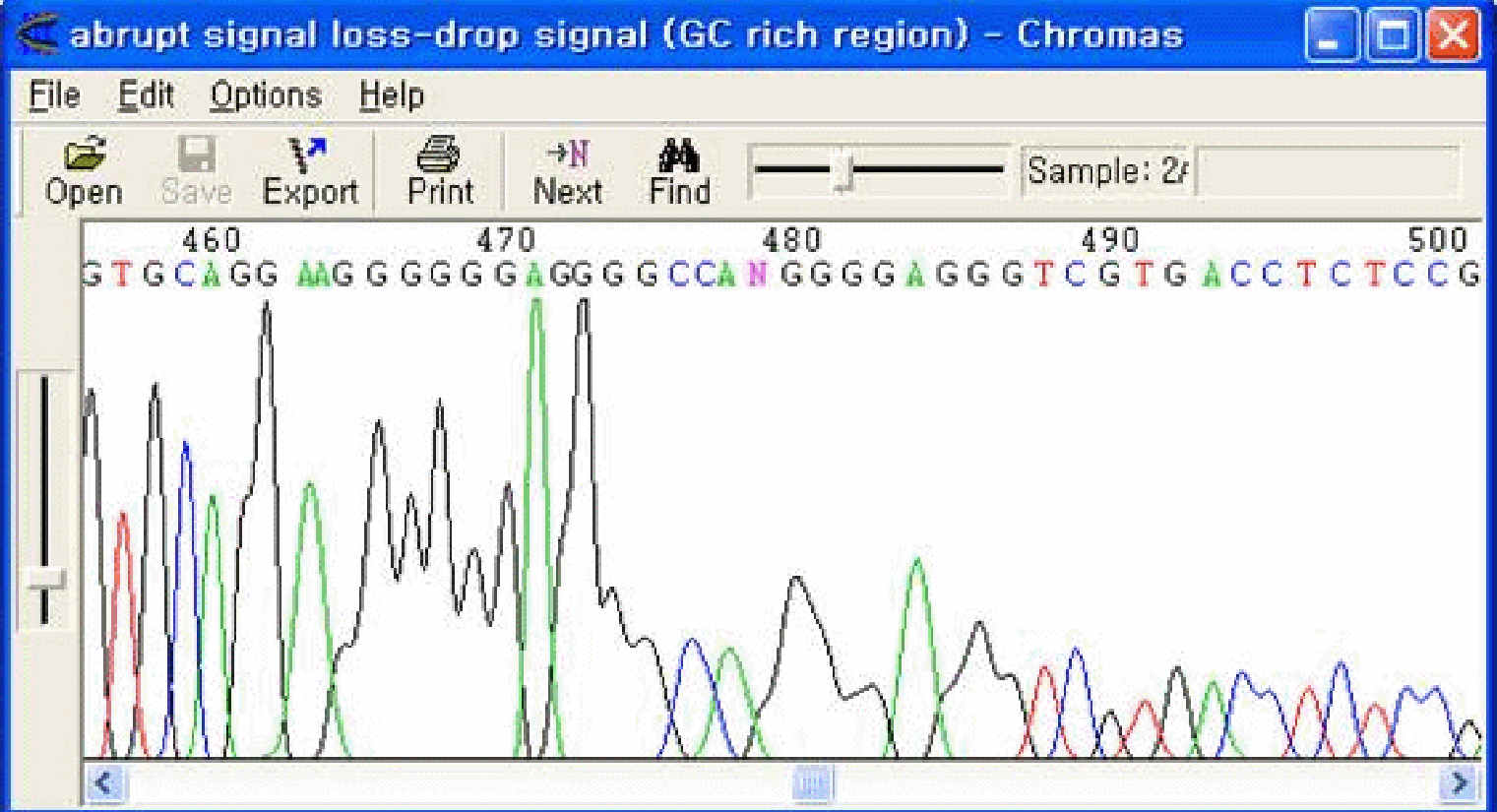
シグナルが急激に減少する現象です。二次構造の形成等により、それ以降のポリマー化が行われず、著しくシグナルが弱くなります。
弊社から納品する解析データは、ab1, txt, pdf, phd, scfの5種類となります。これらのファイルを開くためには専用のソフトウェアが必要となります。下記URLからダウンロードしてください。お客様のご利用し易いファイルにてデータのご確認をお願いいたします。
ABIファイル
専用のソフトウェアが必要となります。下記アドレスより無償ダウンロードできます。
-Chromas Lite (Windows)
http://www.technelysium.com.au/chromas_lite.html
-FinchTV Download
https://digitalworldbiology.com/FinchTV
https://www.thermofisher.com/jp/ja/home/life-science/sequencing/sanger-sequencing/sanger-dna-sequencing/sanger-sequencing-data-analysis.html
PDFファイル
AdobeR ReaderRが必要となります。下記アドレスより無償ダウンロードできます。
http://www.adobe.co.jp/products/acrobat/readstep2.html
PHDファイル
Phredはワシントン大学で開発されたアセンブリに関するソフトウェアです。
シーケンスの波形データを読取り、ベースコールを行い、
各塩基の値を算出します。詳細については http://www.phrap.org をご参照下さい。

 会社説明
会社説明 アカウント作成
アカウント作成
